コミット
633295b0f8
7個のファイルの変更、163行の追加、0行の削除
+ 104
- 0
README.md
ファイルの表示
| @@ -0,0 +1,104 @@ | |||
| # NGS Check mates | |||
| > Author: Ren Luyao | |||
| > | |||
| > E-mail: 18110700050@fudan.edu.cn | |||
| > | |||
| > Git: http://choppy.3steps.cn/renluyao/NGScheckMates.git | |||
| > | |||
| > Last Updates: 2019/02/08 | |||
| ```bash | |||
| source activate choppy | |||
| choppy install renluyao/NGScheckMates | |||
| ``` | |||
| # APP概述 | |||
| NGScheckMates是用来检测某几个测序数据是否来自于同一个人,有以下几种应用场景: | |||
| (1)多组学研究,RNAseq和DNAseq是否是来自同一个人; | |||
| (2)被标注为配对的Tumor和Normal样本是否是来自同一个人; | |||
| (3)同一批样本,多次测序,其中有没有被标错的样本。 | |||
| 推荐直接用fastq模式,优点:如果有多种不同测序文件,比如一个项目中有WES和RNAseq,你要研究WES找到的候选突变是否影响了基因表达量的改变,你需要检查WES和RNAseq的数据是否来自同一个人,以确保分析结果的正确性。直接用fastq模式可以不用单独对RNAseq call germline mutation,以节省时间。这一步将单独运行一个脚本。 | |||
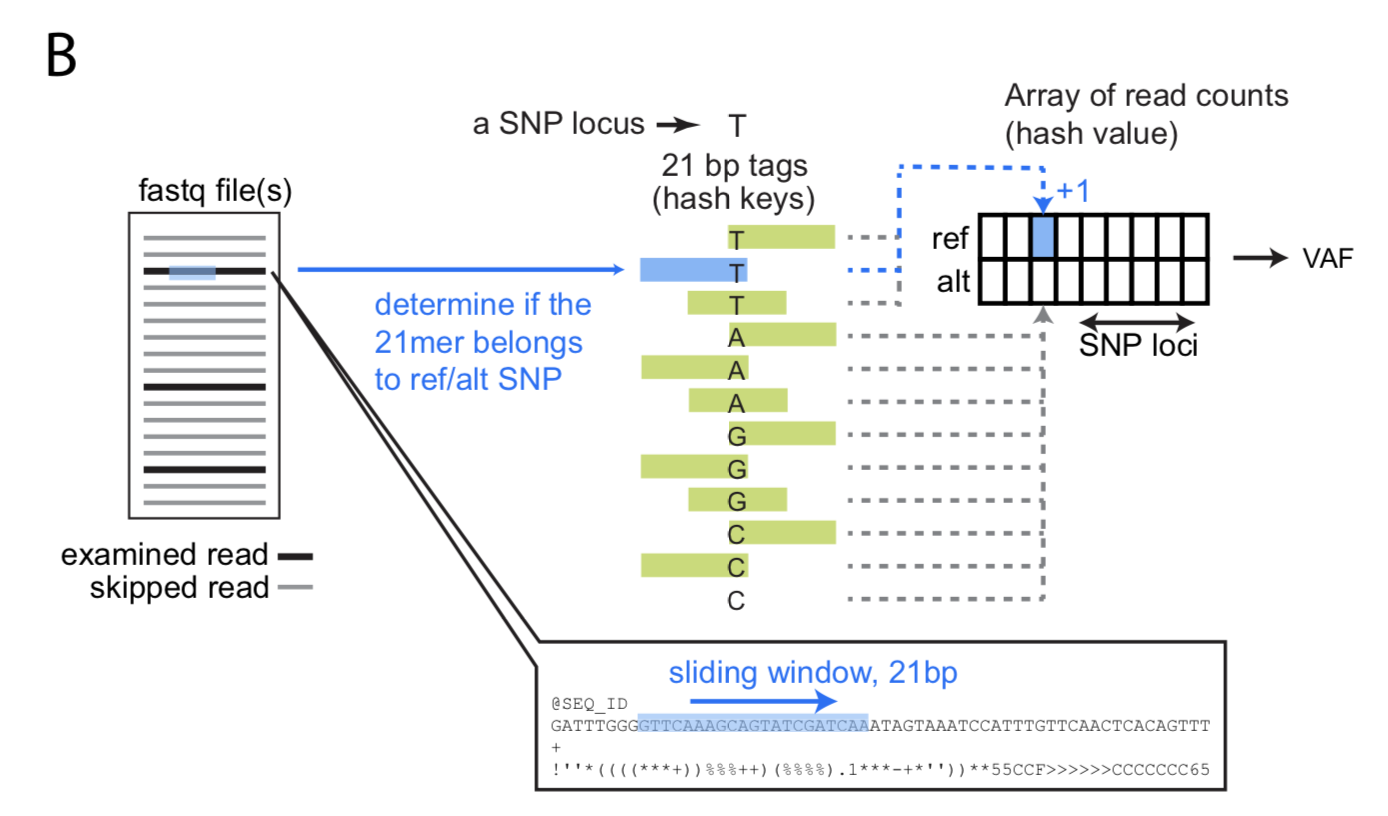
| 基于fastq文件检查样本的配对情况的原理是:他们首先从dbSNP数据库中选择了21067个位于外显子上的SNP用于预测样本配对。对于不用比对的fastq模式,他们在参考基因组中寻找了可以与参考基因组完全匹配的21bp长度的k-mer,位于这些k-mer上的SNP只剩下了11696个。然后用k-mer扫描fastq文件,计算每个SNP的VAF,再根据多个SNP的VAF计算样本间两两的相关性判断两个样本是否来源于一个人。 | |||
|  | |||
| # 流程与参数 | |||
| - Required arguments | |||
| `-l` 需要检测的fastq或者fastq.gz文件的表格,格式如下: | |||
| ```bash | |||
| FASTQ_FILE1 (tab) FASTQ_FILE2 (tab) SAMPLE_NAME (\n) | |||
| Example: | |||
| /data/LSJ_R1.fastq /data/LSJ_R2.fastq LSJ | |||
| /data/LSH_R1.fastq /data/LSH_R2.fastq LSH | |||
| ``` | |||
| `-pt` 是一个包含SNP位点的二进制文件,这些位点可以用与样本的配对检查,在下载包中,路径为`SNP/SNP.pt` | |||
| `-O` 输出文件夹 | |||
| - Optional arguments | |||
| `-N` 输出文件夹的前缀,default:“output” | |||
| `-f` 当你的样本中有父母与孩子或者兄弟姐妹时,加上这个参数,使用更严格的VAF相关系数的阈值 | |||
| `-nz` Use the mean of non-zero depths across the SNPs as reference depth, default: Use the mean depth across all the SNPs | |||
| `-s` The read subsampling rate, default: 1.0 | |||
| `-d` The target depth for read subsampling. NGSCheckMate calculates a subsampling rate based on this target depth. | |||
| `-R` The length of the genomic region with read mapping (default: 3x10^9) used to compute subsampling rate. If your data is NOT human WGS and you use the -d option, it is highly recommended that specify this value. For instance, if your data is human RNA-seq, the genomic length with read mapping is ~3% of the human genome (1x10^8) | |||
| 注意:如果你的fastq文件特别大,它的计算会特别慢,三个参数的使用可以通过subsampling的方法加快运算速度,文献中报道只要0.5X深度的数据就能有很好的预测效果,有两种选择: | |||
| - 只使用 -s ,意思是fastq原始文件的百分之多少,如果你的fastq文件太大,运算速度很比较慢,可以使用其中一部分数据运算,不会影响运算结果,比如,30%就是`-s 0.3` | |||
| - 需要通过使用-d 和-R | |||
| `-L` The length of the flanking sequence of the SNPs, default: 21bp. It is not recommended that you change this value unless you create your own pattern file (.pt) with a different length. | |||
| `-p` 线程数,default:1 | |||
| # APP输入变量与输入文件 | |||
| (1)准备样本文件 | |||
| ```bash | |||
| choppy samples NGScheckMates --output samples | |||
| ``` | |||
| samples文件中输入是 | |||
| - fastq_dir | |||
| fastq文件的地址,阿里云上的地址;如果需要使用多个项目的fastq文件,输入两个项目文件夹的上一级共同目录 | |||
| - Input_file | |||
| 一个txt文件,需要进行计算的文件的详细文件名,文件的地址按照要求修改 | |||
| ```bash | |||
| #read1 #read2 #sample_name | |||
| /cromwell_inputs/*/directory_name/read1.fastq.gz /cromwell_inputs/*/directory_name/read2.fastq.gz sample_name | |||
| ``` | |||
| # APP输出结果 | |||
| # 结果展示与解读 | |||
+ 5
- 0
defaults
ファイルの表示
| @@ -0,0 +1,5 @@ | |||
| { | |||
| "docker": "registry.cn-shanghai.aliyuncs.com/pgx-docker-registry/ngscheckmate:v1.0.0", | |||
| "disk_size": "100", | |||
| "cluster_config": "OnDemand bcs.a2.3xlarge img-ubuntu-vpc" | |||
| } | |||
+ 7
- 0
inputs
ファイルの表示
| @@ -0,0 +1,7 @@ | |||
| { | |||
| "{{ project_name }}.docker": "{{ docker }}", | |||
| "{{ project_name }}.input_file": "{{ input_file }}", | |||
| "{{ project_name }}.fastq_dir": "{{ fastq_dir }}", | |||
| "{{ project_name }}.disk_size": "{{ disk_size }}", | |||
| "{{ project_name }}.cluster_config": "{{ cluster_config }}" | |||
| } | |||
バイナリ
picture/NGSMateCheck.png
ファイルの表示
バイナリ
tasks/.DS_Store
ファイルの表示
+ 29
- 0
tasks/NGScheckMates.wdl
ファイルの表示
| @@ -0,0 +1,29 @@ | |||
| task NGScheckMates { | |||
| File fastq_dir | |||
| File input_file | |||
| String docker | |||
| String cluster_config | |||
| String disk_size | |||
| command <<< | |||
| set -o pipefail | |||
| set -e | |||
| nt=$(nproc) | |||
| export NCM_HOME=/opt/NGSCheckMate | |||
| python /opt/NGSCheckMate/ncm_fastq.py -l ${input_file} -pt /opt/NGSCheckMate/SNP/SNP.pt -O '.' -p $nt -f -s 0.3 | |||
| >>> | |||
| runtime { | |||
| docker:docker | |||
| cluster:cluster_config | |||
| systemDisk:"cloud_ssd 40" | |||
| dataDisk:"cloud_ssd " + disk_size + " /cromwell_root/" | |||
| } | |||
| output { | |||
| File all_txt="output_all.txt" | |||
| File ncm="wd.txt" | |||
| File cor_txt="output_corr_matrix.txt" | |||
| File matched_txt="output_matched.txt" | |||
| File r_script="r_script.r" | |||
| } | |||
| } | |||
+ 18
- 0
workflow.wdl
ファイルの表示
| @@ -0,0 +1,18 @@ | |||
| import "./tasks/NGScheckMates.wdl" as NGScheckMates | |||
| workflow {{ project_name }} { | |||
| File fastq_dir | |||
| File input_file | |||
| String docker | |||
| String cluster_config | |||
| String disk_size | |||
| call NGScheckMates.NGScheckMates as NGScheckMates { | |||
| input: | |||
| fastq_dir=fastq_dir, | |||
| input_file=input_file, | |||
| docker=docker, | |||
| disk_size=disk_size, | |||
| cluster_config=cluster_config | |||
| } | |||
| } | |||
読み込み中…