|
|
5年前 | |
|---|---|---|
| codescripts | 5年前 | |
| pictures | 5年前 | |
| tasks | 5年前 | |
| README.md | 5年前 | |
| inputs | 5年前 | |
| workflow.wdl | 5年前 | |
README.md
WGS-germline Small Variants Quality Control Pipeline
Author: Run Luyao
E-mail:18110700050@fudan.edu.cn
Git: http://choppy.3steps.cn/renluyao/WGS_germline_datapotal.git
Last Updates: 2020/0705
安装指南
# 激活choppy环境
source activate choppy
# 安装app
choppy install renluyao/WGS_germline_datapotal
App概述——中华家系1号标准物质介绍
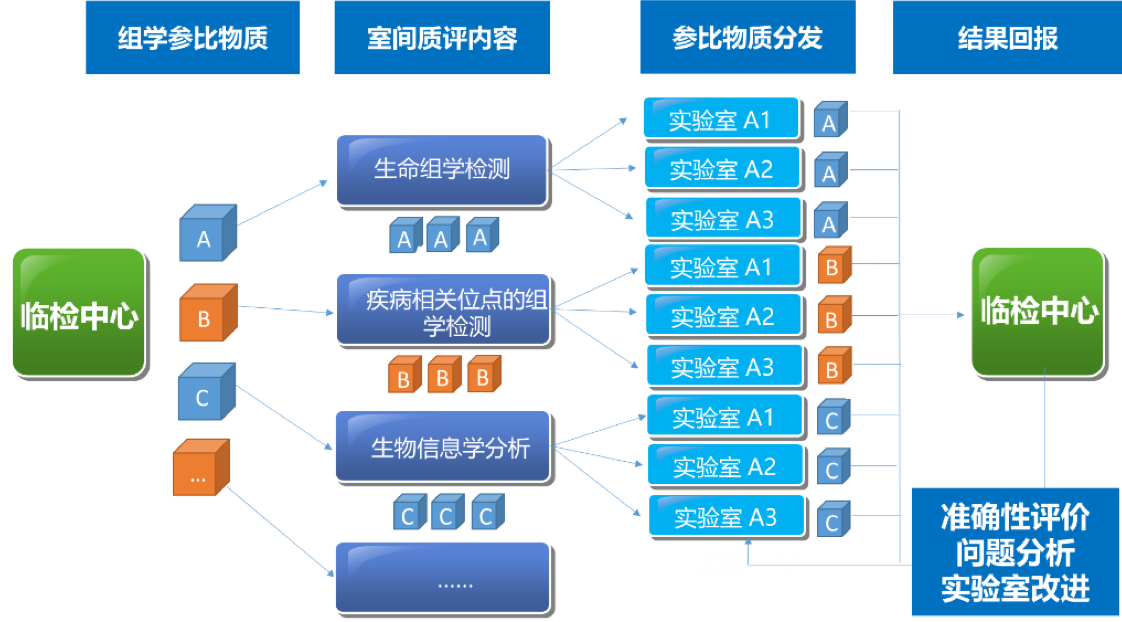
建立高通量全基因组测序的生物计量和质量控制关键技术体系,是保障测序数据跨技术平台、跨实验室可比较、相关研究结果可重复、数据可共享的重要关键共性技术。建立国家基因组标准物质和基准数据集,突破基因组学的生物计量技术,是将测序技术转化成临床应用的重要环节与必经之路,目前国际上尚属空白。中国计量科学研究院与复旦大学、复旦大学泰州健康科学研究院共同研制了人源中华家系1号基因组标准物质(Quartet,一套4个样本,编号分别为LCL5,LCL6,LCL7,LCL8,其中LCL5和LCL6为同卵双胞胎女儿,LCL7为父亲,LCL8为母亲),以及相应的全基因组测序序列基准数据集(“量值”),为衡量基因序列检测准确与否提供一把“标尺”,成为保障基因测序数据可靠性的国家基准。人源中华家系1号基因组标准物质来源于泰州队列同卵双生双胞胎家庭,从遗传结构上体现了我国南北交界的人群结构特征,同时家系的设计也为“量值”的确定提供了遗传学依据。
中华家系1号DNA标准物质的Small Variants标称值包括高置信单核苷酸变异信息、高置信短插入缺失变异信息和高置信参考基因组区。该系列标准物质可以用于评估基因组测序的性能,包括全基因组测序、全外显子测序、靶向测序,如基因捕获测序;还可用于评估测序过程和数据分析过程中对SNV和InDel检出的真阳性、假阳性、真阴性和假阴性水平,为基因组测序技术平台、实验室、相关产品的质量控制与性能验证提供标准物质和标准数据。此外,我们还可根据中华家系1号的生物遗传关系计算同卵双胞胎检测突变的一致性和符合四口之家遗传规律的一致率估计测序错误的比例,评估数据产生和分析的质量好坏。
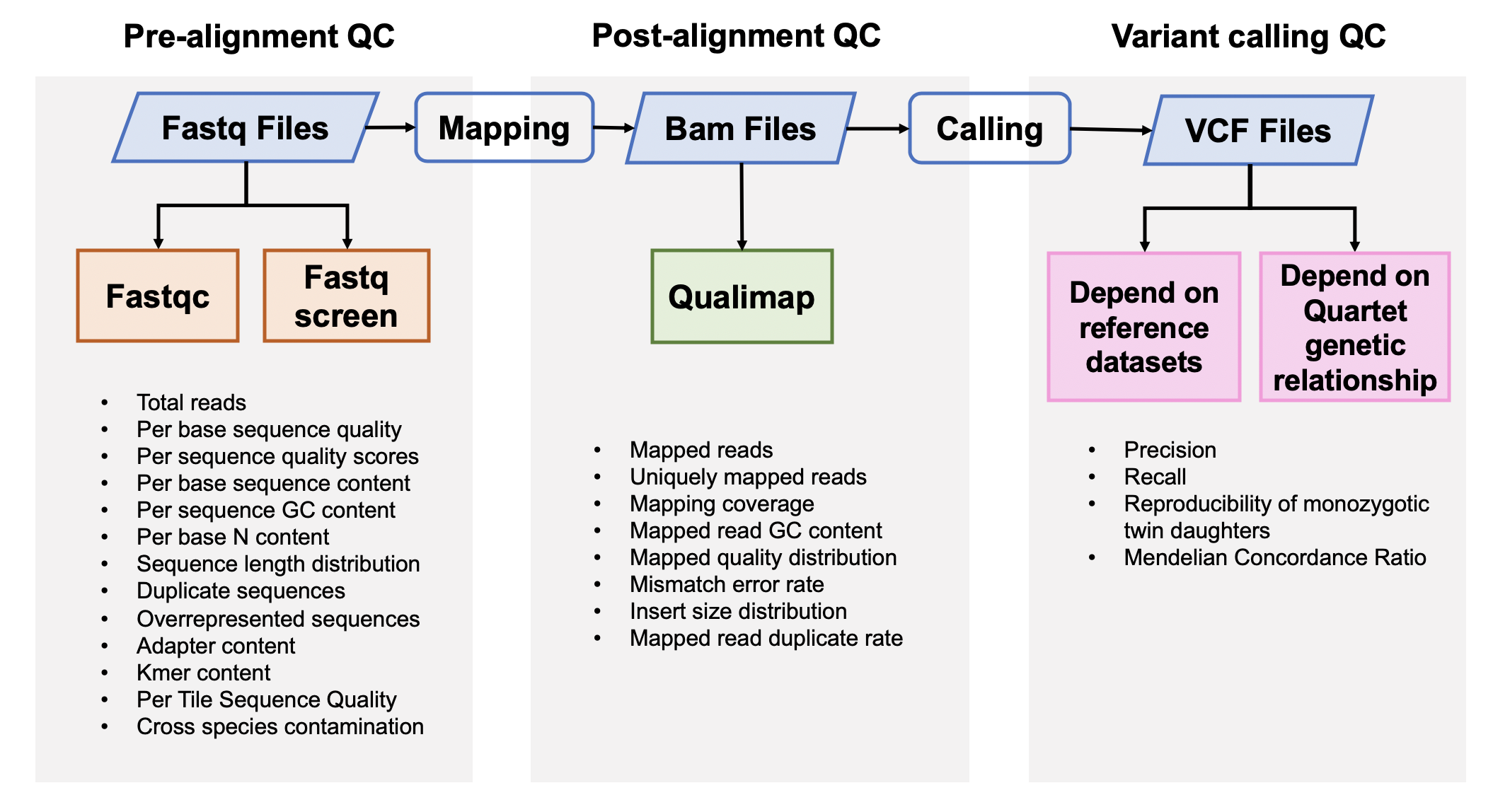
该Quality_control APP用于全基因组测序(whole-genome sequencing,WGS)数据的质量评估,包括原始数据质控、比对数据质控和突变检出数据质控。
流程与参数
1. 原始数据质量控制
Fastqc v0.11.5
FastQC是一个常用的测序原始数据的质控软件,主要包括12个模块,具体请参考Fastqc模块详情。
fastqc -t <threads> -o <output_directory> <fastq_file>
Fastq Screen 0.12.0
Fastq Screen是检测测序原始数据中是否引⼊入其他物种,或是接头引物等污染,⽐比如,如果测序样本 是⼈人类,我们期望99%以上的reads匹配到⼈人类基因组,10%左右的reads匹配到与⼈人类基因组同源性 较⾼高的⼩小⿏鼠上。如果有过多的reads匹配到Ecoli或者Yeast,要考虑是否在培养细胞的时候细胞系被污 染,或者建库时⽂文库被污染。
fastq_screen --aligner <aligner> --conf <config_file> --top <number_of_reads> --threads <threads> <fastq_file>
--conf conifg 文件主要输入了多个物种的fasta文件地址,可根据自己自己的需求下载其他物种的fasta文件加入分析
--top一般不需要对整个fastq文件进行检索,取前100000行
2. 比对后数据质量控制
Qualimap 2.0.0
Qualimap是一个比对指控软件,包含Picard的MarkDuplicates的结果和sentieon中metrics的质控结果。
qualimap bamqc -bam <bam_file> -outformat PDF:HTML -nt <threads> -outdir <output_directory> --java-mem-size=32G
3. 突变检出数据质量控制
突变质量控制的流程如下
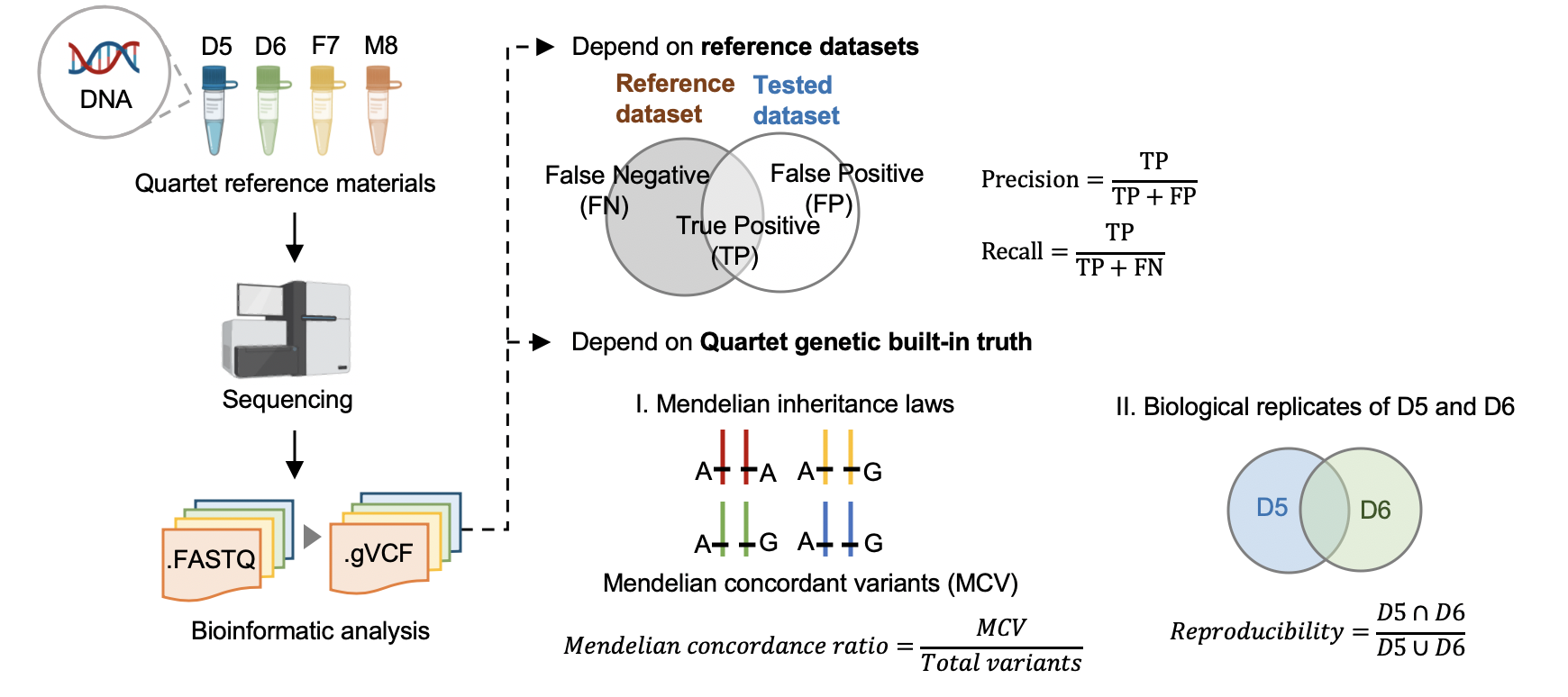
3.1 根据标准数据集的数据质量控制
Hap.py v0.3.9
hap.py是将被检测vcf结果与benchmarking对比,计算precision和recall的软件,它考虑了vcf中突变表示形式的多样性,进行了归一化。
hap.py <truth_vcf> <query_vcf> -f <bed_file> --threads <threads> -o <output_filename>
3.2 根据Quartet四口之家遗传规律的质量控制
Reproducibility (in-house python script)
标准数据集是根据我们整合多个平台方法,过滤不可重复检测、不符合孟德尔遗传规律的假阳性的突变。它可以评估数据产生和分析方法的相对好坏,但是具有一定的局限性,因为它排除掉了很多难测的基因组区域。我们可以通过比较同卵双胞胎突变检测的一致性对全基因组范围进行评估。
Mendelian Concordance Ratio (vbt v1.1)
我们首先将四口之家拆分成两个三口之家进行孟德尔遗传的分析。当一个突变符合姐妹一致,且与父母符合孟德尔遗传规律,则认为是符合Quartet四口之家的孟德尔遗传规律。孟德尔符合率是指四个标准检测出的所有突变中满足孟德尔遗传规律的比例。
vbt mendelian -ref <fasta_file> -mother <family_merged_vcf> -father <family_merged_vcf> -child <family_merged_vcf> -pedigree <ped_file> -outDir <output_directory> -out-prefix <output_directory_prefix> --output-violation-regions -thread-count <threads>
App输出文件
本计算会产生大量的中间结果,这里至说明最后整合好的结果文件
- 原始数据质量控制
- 比对后数据质量控制
- 突变检出数据质量控制