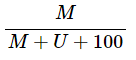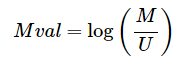|
|
5年前 | |
|---|---|---|
| codescript | 5年前 | |
| picture | 5年前 | |
| tasks | 5年前 | |
| inputs | 5年前 | |
| readme.md | 5年前 | |
| workflow.wdl | 5年前 | |
readme.md
Illumina 850K(EPIC)分析APP
安装指南
#激活choppy环境
'#sourse activate choppy'
#安装APP
choppy install XXXX
APP介绍
甲基化原理简述
Illumina 850K甲基化芯片可同时检测>850,000个位点,覆盖>95%的CpG岛,99%的RefSeq基因,已经成为精准医学研究的重要方法之一。
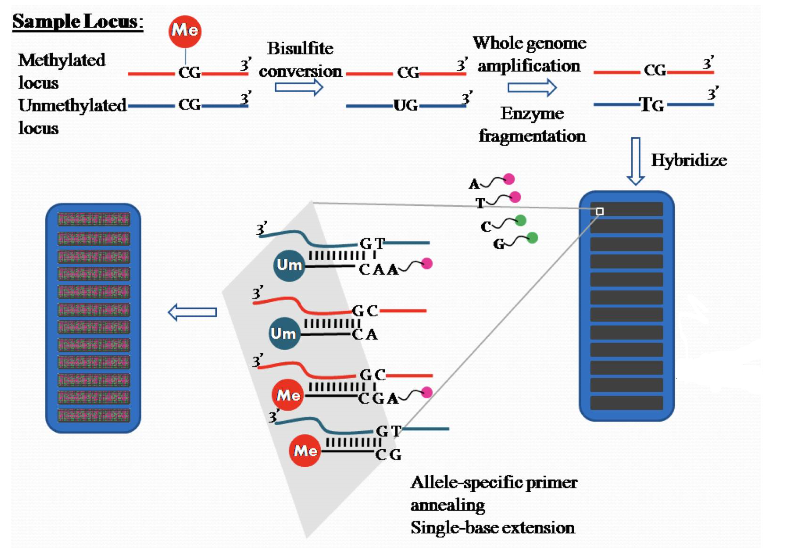
850K芯片采用了两种探针Infinium Ⅰ 和Infinium Ⅱ对样品甲基化进行测定,Infinium I采用了两种bead(甲基化M和非甲基化U,如图显示),而II只有一种bead(即甲基化和非甲基化在一起),这也导致了它们在后续荧光探测的不同,而根据不同探针的bead的荧光值,就可以得到样品各个位点上的甲基化水平。
对于位点甲基化水平的定量测量通常使用两种指标:β值和M值
(其中M代表甲基化信号,U代表非甲基化信号)
β值是最常用的甲基化水平的定量方式,可以直观地了解不同位点的甲基化水平;而M值具有更好的统计学特性,更适用于对样品数据的统计分析。
APP简介
为了更好更便捷的分析全基因组甲基化数据,我们选用了分析850K芯片的R包——minfi包,构建了分析pipeline,可以得到全基因组的各个位点甲基化表达谱。
流程和参数
850K array分析流程
本APP将EPIC芯片的原始数据读入后,过滤了p<0.05的列和p<0.01的行后使用Funnorm方法对数据进行归一化,再过滤其中的SNP位点和性染色体位点,最终得到四张表格(详见输出部分)
使用方法
按照上述步骤安装成功之后,就可以通过下面的命令使用APP
$ Rscript EPIC.modified.R -p xxx -i xxx
具体参数要求见输入模块
输入和输出
输入
需要一个文件夹,其中包含:
▪ idat文件:样本的芯片测序结果文件,命名方式为:“’SentrixID’‘Sentrix_Position’__Grn/Red.idat”
▪ sample_sheet文件:样本的注释信息文件,命名为“sample_sheet.csv”,其中包含Sample_Name,Sentrix_ID, Sentrix_Position, Sample_Group等注释信息
输出
任务完成结束后,便可以在阿里云相应的OSS端生成相应的文件,该输出包含四张含有所有样本M值和beta值的表格(行名为各位点标号,列名为各样本名)。
▪ raw.mVal.txt(原始数据(文件读入后直接得到)的M值)
▪ raw.bVal.txt(原始数据的β值)
▪ filter.p.sex.snp.mVal.txt(过滤和归一化(过滤了低质量位点、SNP位点和性染色体位点,使用Funnorm进行归一化)后数据的M值)
▪ filter.p.sex.snp.bVal.txt(过滤和归一化后数据的β值)